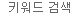General information
실험 계획
성공적인 In-Fusion
® Cloning 반응을 위해서는 선형화 된 vector와 insert 말단의 15 bp 상동 서열이 필요하며, 만약 multiple insert의 삽입을 계획한다면 인접한 insert 혹은 vector 말단의 20 bp 상동 서열을 추천합니다.
- 상동 서열은 PCR 증폭 혹은 oligo 합성을 통해 부가할 수 있다.
- 상동 서열이 12 nt 보다 짧거나 21 nt 보다 긴 것은 추천하지 않는다.
- Translational reading frame은 gene-specific 서열과 vector 상동 서열 사이에 염기를 추가함으로써 조절할 수 있습니다.
- 상동 서열은 반드시 인접한 DNA 조각의 말단에 위치해야 하며, 그렇지 않은 경우 In-Fusion® Cloning에 의해 결합되지 않습니다.
In-Fusion
® 효소는 3’ exonuclease 활성을 가지며, 선형화 된 vector와 insert의 말단을 single strand의 5’ overhang으로 형성합니다. 형성된 overhang이 상보적으로 annealing하며,
E. coli 내에서 circular 형태로 결합됩니다.
- 최소 108 cfu/㎍ supercoiled DNA 이상의 transformation efficiency를 보이는 bacteria strain과 사용하는 것을 추천합니다.
- In-Fusion® Cloning은 선형의 DNA 분자 간의 공유 결합을 이용하지 않습니다.
In-Fusion® 제품의 선택
In-Fusion
® Snap Assembly 제품은 master mix 형태와 동결 건조 형태의 제품 중에 선택할 수 있습니다. 각 제품은 시약과 control 실험을 위한 선형화 된 vector와 2 kb 크기의 insert를 모두 포함하고 있습니다.
Vectors
In-Fusion® Cloning과 호환 가능한 vector
크기나 구성, 제한효소 자리와 관계없이 어떤 vector에도 In-Fusion® Cloning을 적용할 수 있습니다. Cloning 반응은 E. coli에서 circular 형태로 recombinant construct를 형성하나, circular 형태의 vector는 그 자체로 반응에 사용될 수 없습니다. 또한, In-Fusion® Cloning은 선형화 된 DNA 분자 간의 공유 결합을 이용하지 않습니다.
Vector의 크기
In-Fusion® Cloning은 transfer/shuttle vector를 거치지 않고 single reaction만으로 single 혹은 multiple DNA 분자를 large vector (예 - 32.6 ~ 36 kb 크기의 adenoviral vector, 46 kb의 cosmid vector 등)에 삽입할 수 있습니다.
※ In-Fusion® Cloning된 46 kb의 cosmid vector는 competent cell에서 화학적으로 transformation 되었으며, 실제 cosmid packaging reaction에 사용되지는 않았습니다.
Vector 선형화 및 정제
모든 선형화 된 vector는 In-Fusion
® Cloning에 적용 가능합니다. Vector의 선형화는 아래의 방법으로 진행할 수 있습니다.
- 하나 혹은 그 이상의 제한효소 처리
- 효과적인 In-Fusion® Cloning을 위해서는, vector의 말단이 선형화 된 채로 잘 유지되는 것이 중요합니다. 따라서, 높은 효율의 제한효소를 충분한 시간으로 반응시킬 것을 추천합니다. 단, overnight의 반응 시간은 추천하지 않습니다.
- Vector 말단의 dephosphorylation은 필요하지 않습니다. Vector 양 말단의 15 bp 서열이 상보적이지 않은 이상, In-Fusion® 반응으로 vector가 recircularize되지 않습니다.
- 제한 효소를 이용해 선형화 한 vector는 반드시 agarose gel에서 정제해야 합니다 (이 때, DNA 손상을 방지하기 위해, 알루미늄 호일로 커버를 씌워줍니다). 전기영동은 선형화 된 vector와 잘리지 않은 circular 형태의 vector와 구분할 수 있도록 반드시 낮은 전압에서 진행합니다.
- 원하는 cloning site로부터 inverse PCR
- 제한 효소를 사용하지 않기 때문에, 원하는 어느 위치라도 선형화가 가능합니다.
- PCR-mediated mutagenesis (deletion, insertion, base change)를 한 번에 적용할 수 있습니다.
- Insert와의 15 bp의 상동 서열을 vector에 부가할 수 있습니다.
- Inverse PCR 과정에서 vector 서열에 오류가 발생하지 않도록, PrimeSTAR® Max DNA Polymerase (Code R045A)와 같이 높은 proofreading 효율을 가지는 PCR polymerase를 사용할 것을 추천합니다.
- Inverse PCR을 통해 선형화 된 vector는 Cloning Enhancer (Code 639615)를 처리할 수 있으며, 목적 산물 외 발생한 PCR 산물이 존재한다면 Cloning Enhancer (Code 639615)와 gel extraction 과정을 함께 진행하는 것을 추천합니다.
Inserts
In-Fusion® Cloning에 사용될 수 있는 insert
- PCR 증폭 산물 - 인접한 insert DNA, synthetic oligo 혹은 선형화 된 vector의 말단의 상동 서열 포함
- 하나 혹은 그 이상의 제한효소가 처리된 DNA - 인접한 insert DNA, synthetic oligo 혹은 선형화 된 vector의 말단의 상동서열을 반드시 포함
- 50 bp 이상의 Synthetic oligonucleotide
- 양 말단에 인접한 DNA 혹은 선형화 된 vector의 상동 서열 15 bp를 포함
- 반드시 고품질의 정제된 non-phosphorylated된 oligonucleotide를 사용해 반드시 고품질의 oligonucleotide여야 하며, gel 혹은 HPLC를 이용한 정제는 요구되지 않습니다.
Large insert
이 기술은 large fragment의 cloning에 최적화 되어 있습니다. pUC19 vector에 15 kb의 DNA가 insert로 In-Fusion
® Cloning 반응으로 cloning 되었음을 확인하였습니다.
Small insert
150 bp 이하의 작은 insert는 seamless cloning에 적합하지 않을 수 있습니다. Exonuclease activity가 양 말단의 15 ~ 30 bp를 제거할 수 있으므로, insert sequence의 많은 비율을 차지하여 영향을 받을 수 있습니다.
150 bp보다 작은 insert의 경우, ligase를 이용한 전통적인 방식의 cloning을 추천합니다.
Multiple inserts
한 번의 반응으로 5개의 insert를 이용한 multiple-fragment cloning을 성공한 사례가 있습니다.

Primer design
In-Fusion® Cloning에 호환되는 PCR primer
PCR을 위한 forward primer (5’-> 3’ sense strand)와 reverse primer (5’-> 3’ antisense strand)는 아래의 가이드라인에 부합해야 합니다.
- 각 primer의 3’ 말단은 gene-specific 서열로, 목적 유전자를 특이적으로 PCR 증폭을 할 수 있도록 18 ~ 25 nt 길이로 디자인한다.
- 각 primer의 5’ 말단은 선형화 된 vector의 혹은 인접한 insert 말단의 15 nt를 상동 서열로 디자인한다. Multiple-insert cloning을 위해서는 상동 서열을 20 nt로 디자인 할 것을 추천한다. 이 때, 상동 서열의 길이가 12 nt보다 짧거나 21 nt 보다 길게 디자인하는 것은 추천하지 않는다. 15 bp의 상동 서열은 꼭 DNA 말단에 위치해야 하며, 그렇지 않은 경우 In-Fusion® Cloning에 의해 결합되지 않는다.
(선택) Translational reading frame 혹은 제한효소 자리를 보전하기 위해서, gene-specific 서열과 15 bp의 상동 서열 사이에 염기를 추가할 수 있다.
Primer design tools
In-Fusion
®을 위한 PCR primer를 디자인할 때에는 사용자 매뉴얼에서 디자인 조건을 확인할 수 있습니다. 다카라바이오는 Google Chrome 혹은 Mozilla Firefox와 호환되는
온라인 툴을 제공하며, 아래의 application에 대한 디자인이 가능합니다.
- Single-fragment cloning
- Multiple-fragment cloning
- Vectors linearized by restriction digest
- Vectors linearized by inverse PCR
- Prelinearized vectors
- Insert-specific primers designed by the user
- Insert-specific primers generated by the Primer Design Tool
다만, 제공되는
온라인 툴에서는 translational reading frame 조절을 위한 조정 기능을 지원하고 있지 않으므로, 목적하는 실험에서 필요한 경우 직접 조절해 주셔야 합니다.
※
in silico 상에서의 최종 산물을 미리 확인하고자 한다면,
SnapGene Viewer를 이용할 수 있습니다.
그 외 다양한 실험을 위한 primer 디자인 조건
제한효소 자리의 보전
Vector의 선형화를 위해 사용한 제한 효소 자리는 gene-specific 서열과 15 bp 상동 서열 사이에 nucleotide를 추가하여 보존할 수 있습니다. 다카라바이오에서 제공하는
온라인 툴을 활용하면 이를 선택하여 PCR primer를 디자인 할 수 있습니다.
Translational reading frames
In-Fusion
® Cloning은 원하는 insert를 cloning vector에 있는 tag (예 - 형광단백질, Myc, HA 등)와 동일한 translational reading frame을 갖도록 seamless cloning이 가능하며, 아래와 같은 조건을 검토할 수 있습니다.
- In-Fusion® PCR primer 내 gene-specific 서열의 길이를 조정한다.
- Gene-specific 서열과 15 bp의 상동 서열 사이에 염기 서열을 추가한다.
다카라바이오에서 제공하는
온라인 툴에서는 translational reading frame 조절을 위한 조정 기능을 지원하고 있지 않으므로, 목적하는 실험에서 요구되는 경우 직접 디자인해 주셔야 합니다.
Inserting external nucleotide sequences
Small tag, Kozak sequence, restriction site 등 small external nucleotide sequence는 15 bp의 상동서열과 gene specific 서열 사이에 추가할 수 있습니다.
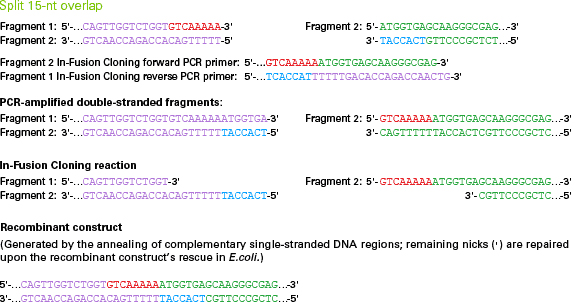
Splitting the 15-nt homologous overlap
15 bp의 상동 서열은 인접한 DNA 간에 나누어 부가하여도 됩니다. 다만, insert와 vector의 상동서열을 나누어 부가하고자 한다면, vector는 inverse PCR을 이용해 선형화 하는 경우에만 적용이 가능합니다.
다카라바이오에서 제공하는
온라인 툴에서는 DNA간 상동 서열을 나누어 부가하는 기능을 지원하고 있지 않으므로, 목적하는 실험에서 요구되는 경우 직접 디자인해 주셔야 합니다.
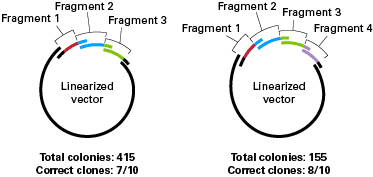
아래 그림은 Fragment 1 (Red)와 Fragment 2 (Blue)의 상동 서열을 나누어 부가한 실험 예시입니다.
Site-directed mutagenesis
In-Fusion
® Cloning은 single 혹은 multiple base change, deletions, insertion이 가능합니다. 보다 자세한 mutagenesis의 정보는 아래의 페이지에서 확인할 수 있습니다.
General guidelines
Molar ratios
In-Fusion
® 효소는 매우 최적화된 효소로 대부분의 실험에서 매우 높은 cloning 효율을 보입니다. Insert와 vector의 일반적인 molar ratio는 제품 매뉴얼 내에서 확인할 수 있습니다.
- 일반적인 single 혹은 multiple fragment cloning 시, insert와 vector의 molar ratio는 2:1로 진행하는 것을 추천한다.
- Multiple fragment를 이용하는 경우, 각 insert는 모두 정제된 선형화 vector와 2:1의 molar ratio로 반응해야 한다. (예 - 2개의 insert를 사용하는 경우, 2:2:1의 molar ratio)
- 가장 작은 크기의 insert가 20 ng이 넘지 않도록 나머지 DNA의 양을 계산하며, 이 때의 molar ratio는 2:1이 되도록 한다.
- 150 ~ 350 bp 크기의 small DNA fragment의 cloning을 위해서는 insert와 vector의 molar ratio는 3 ~ 5:1을 추천한다.
- 50 ~ 150 bp 크기의 short synthetic oligo의 cloning을 위해서는 insert와 vector의 molar ratio는 5 ~ 15:1을 추천한다.
- Non-phosphorylated oligonucleotide도 In-Fusion® Cloning을 적용할 수 있다. 하지만, 본 기술은 3’ exonuclease 활성을 이용하기 때문에 terminal 3’ OH기가 필요하다.
다카라바이오는 molar ratio, insert 길이 (bp), vector 길이 (bp)를 고려한 insert와 vector의 반응 양을 계산할 수 있는
온라인 툴을 제공하고 있습니다.
Control reactions
제품 내 포함되어 있는 pUC19 control vector와 control insert를 이용한 control test를 항상 실험군과 같이 진행할 것을 권장합니다. 만일 실험에서 원하지 않은 결과가 확인될 때에는 control 반응을 통해 어느 과정에서 troubleshooting이 필요한지 확인할 수 있습니다.
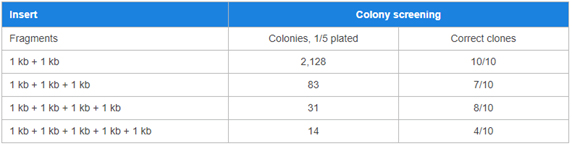
일반적으로 Negative colony는 5% 미만의 blue colony로 나타나며, white colony는 strain에 따라 약간씩 달라질 수 있습니다. 일반적으로 5% 미만의 white colony가 백그라운드로 관찰될 수 있으며, 내부 분석에 따르면 95% 이상의 colony로부터 원하는 결과를 얻었습니다. 총 생성되는 colony의 수와 관계없이 correct colony의 비율이 유지되며, 이는 In-Fusion® 기술이 높은 cloning 효율을 나타냄을 말한다.
Incubation time
반응 시간을 늘리는 것은 추천하지 않습니다. 긴 시간 반응하면, insert와 vector 말단에 생기는 single-strand region이 길게 형성되어, 이로 인해 상동 서열이 잘 annealing 되지 않아 cloning 효율이 오히려 낮아질 수 있습니다.
상동 서열의 위치
15 bp의 상동 서열은 반드시 vector와 insert 양 말단에 정확하게 위치해야 합니다. 15 bp의 상동 서열 vector 상동 서열이 DNA 말단에 반드시 위치해야 하며, 이 점이 준수되지 않는 경우 In-Fusion® 반응이 일어나지 않을 수 있습니다. PCR을 통해 선형화 된 vector의 경우, 제한효소의 자리와 상관없이 원하는 어느 위치라도 cloning할 수 있으며, 원하는 곳 (선형화 된 말단)의 양쪽 15 bp를 상동 서열로 이용하면 됩니다.
상동 서열의 길이
In-Fusion® 반응 시에는 single insert cloning의 경우 15 bp, multiple fragment cloning의 경우 20 bp의 상동 서열을 추천하며, 12 bp 보다 짧거나 21 bp 보다 긴 길이의 상동 서열을 사용하는 것은 추천하지 않습니다.
In-Fusion® PCR primer의 조건
- In-Fusion® PCR primer의 3’ 말단은 gene-specific한 서열로, template 증폭에 사용되기 위해서는 18 ~ 25 bp의 필요하다.
- Primer Tm값을 결정하기 위해서는 PCR primer 분석을 위한 software (예 - IDT technologies사의 OligoAnalyzer 3.1)를 활용할 수 있다. 일반적인 PCR 과정에서 추천하는 Mg2+, Na+, dNTP 농도를 추천한다.
- 최적의 증폭을 위해서, In-Fusion® primer 3’ gene-specific 서열의 annealing 온도를 이용해 초기 3-5 cycle을 수행하고, 남은 cycle은 primer 전체의 annealing Tm값을 이용해 수행한다.
PCR 조건
PCR 증폭 산물은 In-Fusion
® Cloning 반응 전에 반드시 정제되어야 합니다.
※ 대부분의 경우,
Cloning Enhancer (Code 639615)를 처리한 후의 추가적인 column purification혹은 gel extraction 과정은 요구되지 않습니다. 하지만, 보다 좋은 cloning 결과를 얻기 위해서는 PCR로 선형화 된 vector의 경우, 목적 산물 외 발생한 PCR 산물을 제거하기 위해,
Cloning Enhancer (Code 639615)와 gel extraction 과정을 함께 진행하는 것을 추천합니다.
Transformation
호환 가능한 세포
In-Fusion
® Cloning 은 최소 10
8 cfu/㎍ supercoiled DNA 이상의 transformation efficiency를 보이는 bacteria strain과 사용하는 것을 추천합니다.
- Stellar™ Competent Cells (Code 636763)은 일반적인 cloning 목적에 사용될 수 있는 E. coli strain으로, In-Fusion® Cloning과 사용 시 가장 최적의 효율을 보인다.
- Stellar™ Competent Cells (Code 636763)은 large vector (예 - BACs, fosmide)나 반복 서열이 많은 (reiterated sequence) (예 - retroviral/lentiviral vector의 LTR (Long Terminal Repeats) 서열, adenoviral vector의 ITR (Inverted Terminal Repeats) 서열 등)의 cloning에 사용 가능함이 확인 되었다.
- TOP10 세포나 같은 유래의 ccdB Survival 2T1R E.coli, 혹은 DH10B, MC1061 등과 같은 strain을 사용하는 것은 낮은 수의 recombinant clone의 결과로 이어질 수 있다. 만일 multiple fragment cloning 혹은 low copy number의 vector를 사용하는 경우, 특히 결과에 큰 영향을 받을 수 있다.
아래와 같은 bacteria strain은 In-Fusion
® Cloning 반응에 추천하지 않습니다.
- E. coli strains lacking recA1 or endA mutations
- E. coli strains engineered for a particular application (e.g., large-scale protein expression)
- Gram-positive bacterial strains
- Bacterial cells carrying nupG (deoR) mutations
※ 만일 In-Fusion
® Cloning과의 호환성이 확인되지 않은 특정 bacteria strain을 불가피하게 사용해야 하는 경우, 반응 산물을 1:5로 희석하는 것이 transformation efficiency 증가에 도움을 줄 수 있습니다.
최적의 transformation 비율
Transformation 효율에 영향을 줄 수 있는 vector와 insert 특성
일부 vector와 insert는 반복되는 서열 (retroviral 혹은 lentiviral LTRs, adenoviral ITRs 등)을 포함하고 있을 수 있습니다. 이러한 DNA를 적용하는 경우, 재조합 시의 rearrangement를 방지하고 E.coli 내에서의 증폭 과정에서의 안정성을 높이기 위해 bacterial transformation 과정을 최적화하는 것이 필요할 수 있습니다. Heat shock 후, 120 ~ 160 rpm에서 회복시킨 후 transformant를 항생제의 농도나 배양 온도 (예 - 25 ~ 30 °C 등)를 조절하여 plate에서 배양합니다.
Selected applications
목적 유전자를 형광 단백질과 함께 발현시키고자 하는 경우 (easy protocol)
- 1. 형광 단백질을 발현하는 In-Fusion® Ready vectors를 사용한다.
- 2. Vector 정보를 확인한 후, In-Fusion® PCR primer를 디자인한다
※ Translational reading frame 보존을 위해, 형광 단백질 upstream에서 목적 유전자를 발현하는 경우, cloning하는 서열 내에는 STOP codon을 포함하지 않도록 한다. 만약 형광 단백질 downstream에서 목적 유전자를 발현하는 경우, cloning 서열 내에는 STOP codon을 포함해도 되나, 형광 단백질 서열 내에는 포함하지 않도록 한다.
- 3. Step 2에서 디자인한 In-Fusion® PCR primer를 이용해 목적 유전자를 증폭한다.
- 4. 증폭 산물을 정제한다.
- 5. 매뉴얼에 따라, In-Fusion®을 이용한 cloning을 수행한다.
목적 유전자를 형광 단백질과 함께 발현시키고자 하는 경우 (alternative protocol)
- 1. 형광 단백질을 발현하는 원하는 vector를 선택하고, 제한효소나 inverse PCR을 이용해 선형화한다.
- 2. 선형화 된 vector만 얻을 수 있도록 gel extraction으로 정제한다.
- 3. 온라인 툴을 이용하여, In-Fusion® PCR primer를 디자인한다.
- 4. Step 3에서 디자인한 In-Fusion® PCR primer를 이용해 목적 유전자를 증폭한다.
- 5. 증폭 산물을 정제한다.
- 6. 매뉴얼에 따라, In-Fusion®을 이용한 cloning을 수행한다.
Large vector를 이용한 cloning
In-Fusion® 기술은 single 혹은 multiple DNA fragment를 한번의 반응으로 transfer/shuttle vector 없이 바로 large vectors (예 - 32.6 ~ 36 kb의 adenoviral vectors)에 쉽게 cloning 할 수 있습니다.
Large insert를 이용한 cloning
In-Fusion
® 기술은 large fragment의 cloning에 최적화 되어 있으며, pUC19 vector에 15 kb의 DNA가 insert로 In-Fusion
® Cloning 반응으로 cloning 되었음을 확인하였습니다.

Multiple-fragment cloning
한 번의 In-Fusion
® 반응으로 5개의 insert를 이용한 multiple-fragment cloning을 성공한 사례가 있습니다.
Site -directed mutagenesis
In-Fusion
® Cloning은 inverse PCR을 통해 single 혹은 multi base changes, deletion, insertion 등이 가능합니다. 이 application은 실험에 사용하는 PCR 효소의 high proofreading activity 영향이 큽니다. 다카라바이오는
PrimeSTAR® Max DNA Polymerase (Code R045A)의 사용을 추천하며, 이 효소는 최대 6 kb의 human gDNA, 10 kb의
E.coli gDNA, 15 kb의 lambda DNA를 샘플로 증폭이 가능하며, GC-rich template에서도 최소한의 error rate를 보입니다.
- Inverse PCR을 위한 모든 PCR primer는 circular 형태의 vector에서 반대 방향으로 DNA 합성을 하도록 디자인한다.
- Forward, reverse primer의 3’ end는 특이적이고 효과적인 증폭이 가능한 template와 상보적인 서열 18 ~ 25 bp로 구성한다.
- Mutation은 forward, reverse primer의 5’ end에 위치한 상동서열 15 bp 내에 포함되어야 한다. 이 상동 서열은 mutation을 포함하는 vector의 recirculation하는 과정에서 사용된다.
- Single 혹은 multi base changes, deletions, insertions은 한 번의 In-Fusion® 반응으로 도입할 수 있다.
- 긴 길이의 deletion의 경우, deletion하고자 하는 위치를 forward, reverse primer의 3’ end로 하고, 5’ end에 상동 서열을 포함하도록 한다.
- Inverse PCR을 진행하면, 5’, 3’ end가 상동한 linear double-strand 형태의 vector를 얻을 수 있고, 15 bp의 상동 서열은 In-Fusion® 반응 후 E. coli 내에서 수복되게 된다.
- Inverse PCR로 증폭된 vector는 반드시 Cloning Enhancer (Code 639615)를 처리하여, 기존의 vector를 제거한다. 전기영동을 통해 선형화 된 vector로부터 circular 형태의 vector를 제거하는 과정이 추가적으로 필요할 수도 있다.
Cloning shRNA (small hairpin RNA)
50 bp 이상의 shRNA double-stranded DNA oligonucleotide는 In-Fusion
® 기술을 이용해 shRNA 발현 vector에 cloning 할 수 있다.
- 50 ~ 150 bp의 짧은 합성 oligo는 In-Fusion® cloning 시, oligo와 vector의 molar ratio를 5 ~ 15:1의 비율로 적용할 것을 추천한다. Oligo 길이에 따라, 최적의 비율은 달라질 수 있다.
- 고품질의 desalting으로 정제한 non-phosphorylated oligo를 In-Fusion® 반응으로 cloning할 수 있다. 단, In-Fusion® 효소는 3’ exonuclease 활성을 이용하기 때문에 terminal 3’ OH기가 필요하다.
※ siRNA와 같이, 모든 antisense oligonucleotide가 direct cell transfection을 위해 디자인 및 테스트된 것이 아니기에, vector로부터 shRNA가 발현되었을 때 다 동등한 효율을 보이지는 않습니다. 일반적으로, shRNA로 발현시키기 위해서는 목적 서열에서 다양한 방향을 가지는 sense 혹은 antisense strand 형태로 siRNA를 다시 디자인 할 것을 추천합니다.
- 효율적인 knockdown을 위해서는, 일반적으로 최소한 4개 이상의 다른 shRNA construct를 stable cell line 혹은 in vivo 적용 전에 transfection이 용이한 cell line에서 테스트해볼 것을 권장한다.
- Recombinant shRNA vector를 구별하기 위해, diagnostic restriction site (MluI)를 RNA Polymerase III Termination Signal로 shRNA oligo의 downstream에 삽입할 수 있다.
Cloning a microRNA (miRNA) precursor
- 100 ~ 300 bp의 miRNA precursor DNA 서열은 gDNA로부터 증폭하여, miRNA 발현 vector의 형광 단백질 3’ UTR로 cloning 할 수 있다.
- In-Fusion® Cloning을 위해, 100 ~ 300 bp 길이의 miRNA precursor가 PCR 증폭될 수 있도록 miRNA expression vector 말단의 상동 서열 15 bp를 포함해야 한다. Vector는 반드시 형광 단백질의 3’ UTR 부위에서 선형화 되어야 한다. miRNA precursor와 vector의 molar ratio는 3 ~ 5:1의 비율을 추천하며, precursor의 길이에 따라 최적의 비율을 결정하여 반응시킨다.
-
세포 내에서 miRNA precursor는 형광 단백질과 함께 발현 되며, 아래의 특징을 보인다.
- 형광 단백질의 발현은 세포 내 형광 labeling으로 나타난다.
- miRNA precursor은 형광 label된 세포에서의 목적 유전자의 knockdown을 유도한다.