ThruPLEX® 적용 논문
적용 제품 |
적용 논문 |
ThruPLEX® DNA-Seq Kit |
Differential activation mechanisms of two isoforms of Gcr1 transcription factor generated from spliced and un-spliced transcripts in Saccharomyces cerevisiae |
Epigenetic alteration contributes to the transcriptional reprogramming in T-cell prolymphocytic leukemia |
https://doi.org/10.1093/nar/gkaa1221
Differential activation mechanisms of two isoforms of Gcr1 transcription factor generated from spliced and un-spliced transcripts in Saccharomyces cerevisiae
Seungwoo Cha, Chang Pyo Hong, Hyun Ah Kang, Ji-Sook Hahn
아래 내용은 다카라코리아에서 직접 번역한 내용으로, 해석하는 과정에서 원작자의 의도와 상이한 내용이 포함되어 있을 수 있습니다. 학술 정보를 목적으로 하는 경우 원문을 참고하여 주시기를 바랍니다.
ㄴ Abstract 미리보기
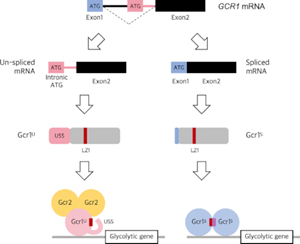
Gcr1은 Saccharomyces cerevisiae의 glycolytic 유전자에 중요하게 관여하는 전사 인자로써, 최근 un-spliced transcript 유래의 Gcr1U와 spliced transcript 유래의 Gcr1S, 두 개의 isoform이 존재하는 것이 밝혀졌다. 본 연구에서는 CRISPR/Cas9를 이용해 Gcr1U 또는 Gcr1S만을 발현하는 균주를 제작하고, isoform에 따라 보이는 activation mechanism의 차이를 설명한다.
Gcr1U monomer는 Gcr2 homodimer와 활성 복합체를 형성하는 반면, Gcr1S는 Gcr2가 존재하지 않을 때 homodimer 형태로 작용한다. 55개의 잔기로 구성된 USS domain은 Gcr1U의 N-terminus에만 존재하며, Gcr1U 뿐 아니라 Gcr1S의 dimerization까지 억제한다. Gcr1S monomer는 에탄올 활용에 필요한 mitochondrial aldehyde dehydrogenase를 인코딩하는 ALD4 유전자 과발현에 의해 회복할 수 있는 ALD4 promoter에 직접 결합하여, 발효에서 호흡으로 대사가 전환하는 것을 억제한다. Gcr1U와 Gcr1S는 유사한 유전자 조절에 관여하지만 성장기에 따라 차등한 활성을 보며, 이는 isoform이 환경 조건에 따른 별도의 활성화 기전을 통해 다른 역할을 하고 있음을 보여준다.
ㄴ Takara 제품을 이용한 method 미리보기
ChIP-seq을 위해 triplicate로 배양된 세포를 각각 회수하여 sonication한 후, 총 3 ㎖ 샘플을 준비했다. 두 세트의 샘플 600 ㎕는 각 protein tag에 따른 항체와 beads를 함께 incubation한 후 reverse crosslinking 하였으며,
이 후 phenol-chloroform-isoamyl alcohol (25:24:1)를 처리하고, -80 °C에서 에탄올과 glycogen으로 침전시켰다. 건조된 pellet은 물로 용해하여 높은 DNA 농도를 위해 모아서 분석하였다.
Gcr1U와 Gcr1S의 ChIP DNA fragment (1-5 ng)의 sequencing library는 Takara의
ThruPLEX® DNA-Seq Kit로 제작하였으며,
제조사의 프로토콜에 따라 실험을 진행하였다. 간략하게, DNA fragment의 end-repair 과정 후 3’ A-tailing, adapter ligation를 진행하고, 15 cycle의 PCR을 통해 DNA를 증폭한 후 정제하였다. 제작된 library는 Illumina
®의 HiSeq2500 기기를
사용해 single-end sequencing, 50 cycle로 분석하였다.
ㄴ 논문에서 사용된 Takara 관련 제품 알아보기
ThruPLEX® DNA-Seq Kit (Code R400674)
- 50 pg ~ 50 ng DNA 샘플로부터 Illumina® 분석용 DNA-seq library를 제작
- FFPE DNA, ChIP DNA 등 극소량 혹은 degradation 된 샘플에도 적용 가능
- Single tube 내 3-step으로 완료되는 매우 간편한 프로토콜
- 동일 목적의 타사 제품들과 달리, 중간 정제과정이 없어 샘플 소실 및 오염 최소화
ThruPLEX
® 기술 도입으로, stem loop adapter를 이용해 background data 영향 최소화
https://doi.org/10.1038/s41598-021-87890-9
Epigenetic alteration contributes to the transcriptional reprogramming in T-cell prolymphocytic leukemia
Shulan Tian, Henan Zhang, Pan Zhang, Michael Kalmbach, Jeong-Heon Lee, Tamas Ordog, Paul J. Hampel, Timothy G. Call, Thomas E. Witzig, Neil E. Kay, Eric W. Klee, Susan L. Slager, Huihuang Yan & Wei Ding
아래 내용은 다카라코리아에서 직접 번역한 내용으로, 해석하는 과정에서 원작자의 의도와 상이한 내용이 포함되어 있을 수 있습니다. 학술 정보를 목적으로 하는 경우 원문을 참고하여 주시기를 바랍니다.
ㄴ Abstract 미리보기
T 세포 전림프구성 백혈병 (T cell prolymphocytic leukemia, T-PLL)은 치명적인 임상 결과를 보이는 희귀 질환으로, 세포유전학적 분석, whole-exome / whole-genome sequencing을 통해 T-PLL이 inversion, translocation, CNV (copy number variation)을
비롯한 주요한 구조 변경을 나타내는 것이 확인되었다. 재발성 체세포 돌연변이는 염색질 조절인자를 코딩하는 유전자와 JAK-STAT 신호 전달 경로에서도 확인되었다. 대다수의 암에서 후성 유전학적 변화가 특징으로 나타나지만, T-PLL에서의
genome-wide epigenomic profiling에 대한 연구가 보고되지 않아, 이의 암 발생 과정에 대한 연구에 제한이 있었다. 따라서, 본 논문의 저자들은 T-PLL이 유발되는 데에 후성 유전학적인 매커니즘이 중요한 역할을 한다고 가정하였다.
이 가설을 체계적으로 확인하기 위해, 본 논문에서는 T-PLL 환자와 건강한 사람을 대상으로 진행한 H3K4me3와 H3K27ac의 ChIP-seq과 RNA-seq 데이터를 이용해 조절 영역에 대한 genome-wide map을 제작하였다. 이를 바탕으로
T-PLL에서 down-regulated된 유전자가 주로 방어 기작, 면역 체계 혹은 적응 면역 반응에 주로 관여하고 있는 반면, up-regulated된 유전자는 발달 과정 및 세포 운명을 결정하는데 주요한 역할을 하는 WNT 신호 전달 경로와 연관이 있다는 것을 발견하였다.
특히, 본 논문에서 분석한 바에 따르면, T-PLL에서 DNA 손상 반응과 T 세포 활성화에 관여하는 유전자, 종양 유전자의 후성 유전학적 조절과 관련된 면역 전사 인자와 결합하는 motif가 매우 상이한 피크를 보이면서 조절 환경에서 전체적인 변경을 보였다.
이를 종합하여 볼 때, 본 논문은 T-PLL의 후성 유전학적인 조절 장애와의 관련성을 증명하였다.
ㄴ Takara 제품을 이용한 method 미리보기
H3K4me3는 주로 promoter에, H3K27ac는 active enhancer에 위치하며, 이 두개의 histone mark는 유전자 조절 영역을 mapping하는 데 널리 사용되고 있다. 따라서, 본 논문의 저자들은 anti-H3K27ac 항체와 anti-H3K4me3 항체를 이용해 ChIP을 수행하였다.
이후 5 ng의 ChIP DNA와 Takara의
ThruPLEX® DNA-Seq Kit를 이용해 ChIP-seq library를 제작하였다. ChIP enrichment는 real-time PCR을 이용해 검증하였으며,
제작된 library는 양 말단으로부터 51개 혹은 101개 염기로부터 HiSeq4000 플랫폼을 이용해 분석되었다.
ㄴ 논문에서 사용된 Takara 관련 제품 알아보기
ThruPLEX® DNA-Seq Kit (Code R400674)